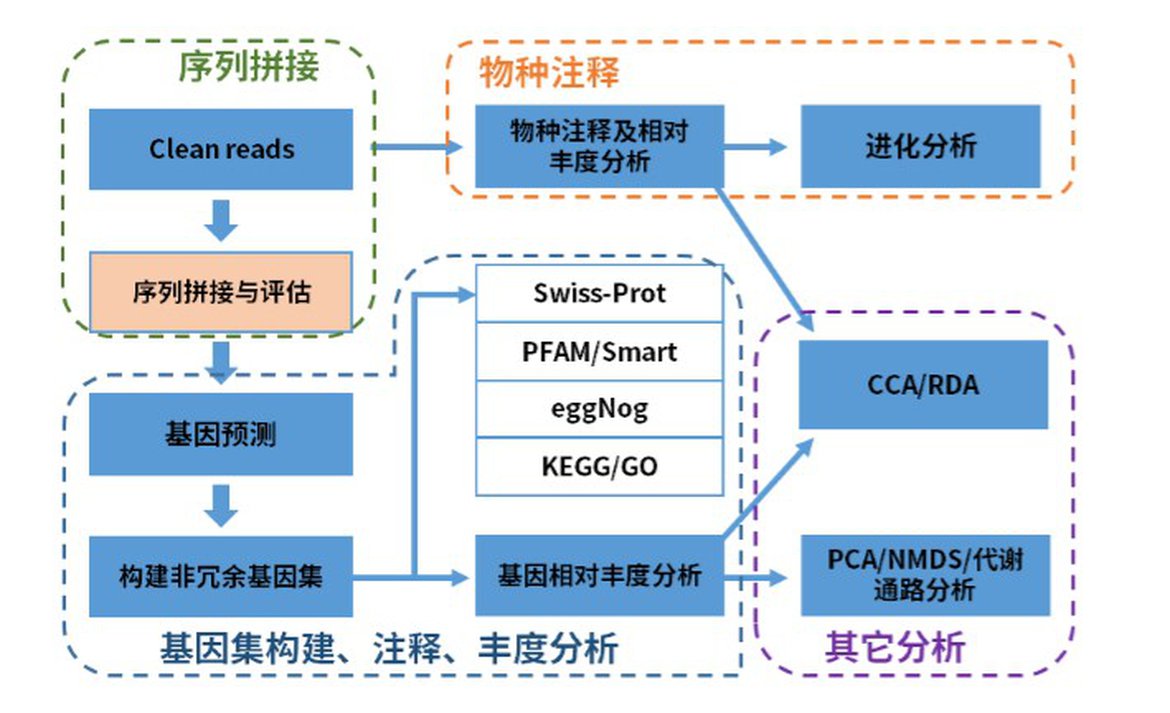
宏基因组学(也叫环境基因组学),是采用高通量测序技术,以获得特定生境微生物群落的全部或部分基因信息为目的,进而研究环境微生物的基因功能、代谢网络、物种丰度、系统进化和群落结构等。
我们通过对环境样本提取总DNA、建库和测序,通过生物信息学方法,进行宏基因组拼接、基因预测和注释、基因丰度和物种丰度分析、PCoA分析和NMDS分析、CCA/RDA分析等,探索不同生境的基因丰度、代谢途径、物种丰度等的差异。
主要分析:环境样本
测序平台:Illumina HiSeq Xten (小片段PCR-free文库,~450bp);
PacBio Sequel (~20kb文库);
分析流程
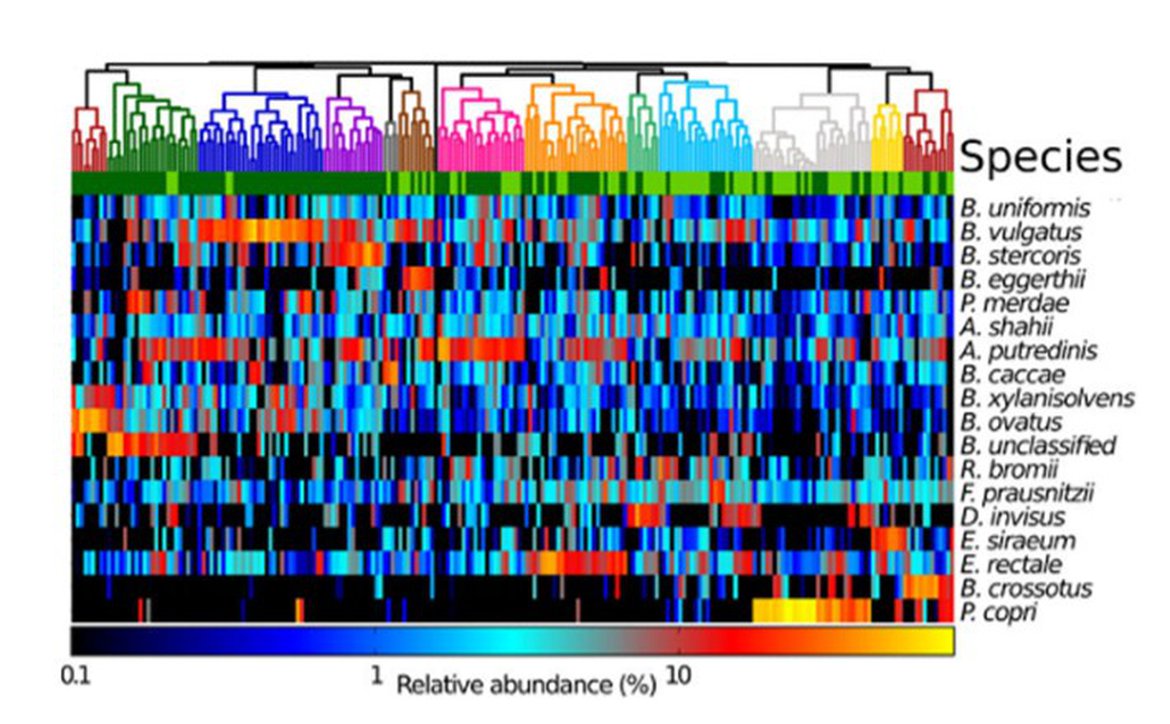
例图1、组内样本具有相似的菌群组成
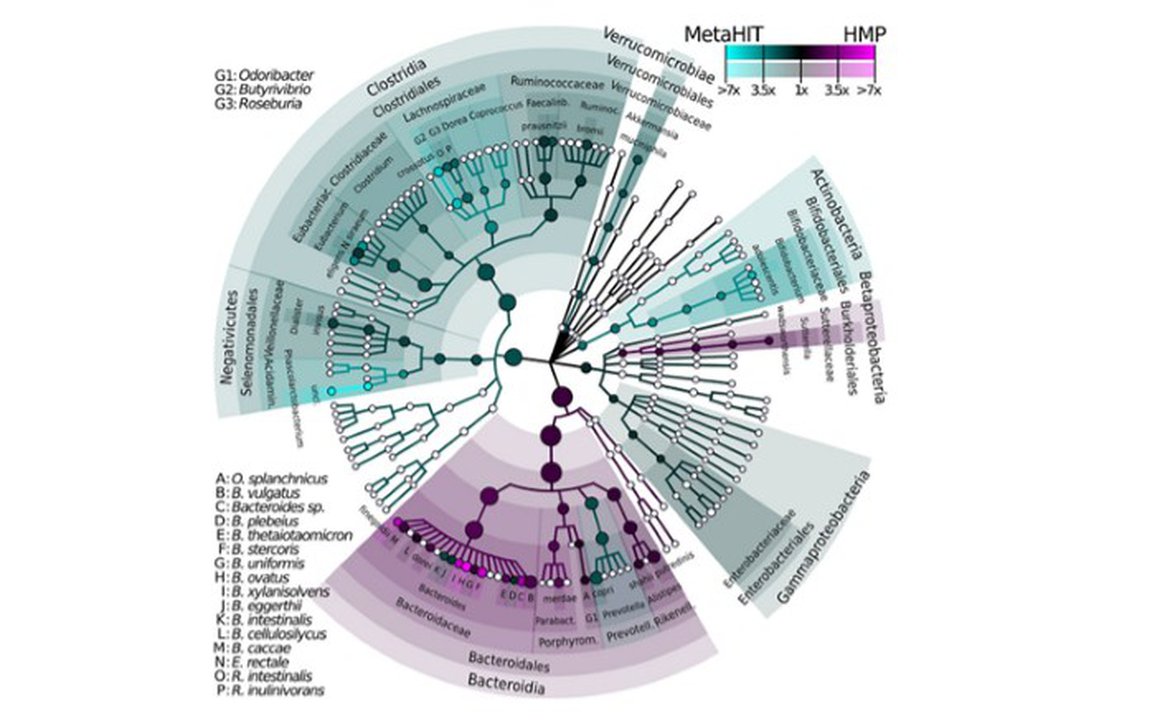
例图2、菌群进化分支图
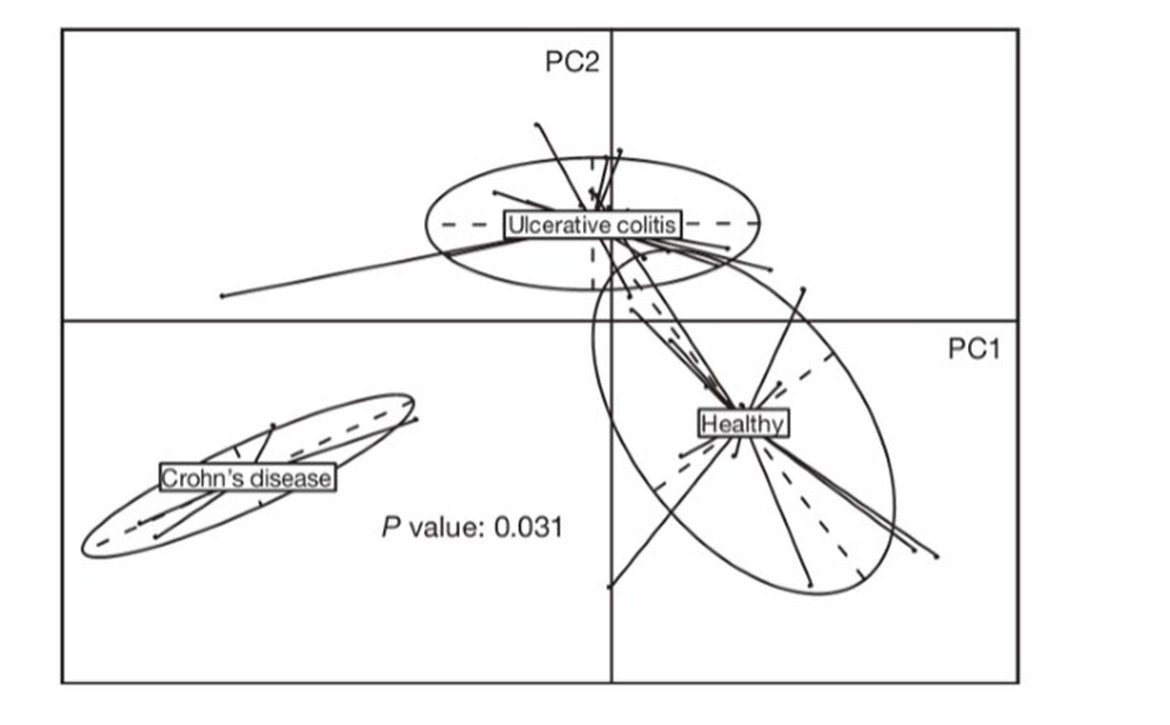
例图3、PCA显示疾病组与健康组的菌群丰度差异明显
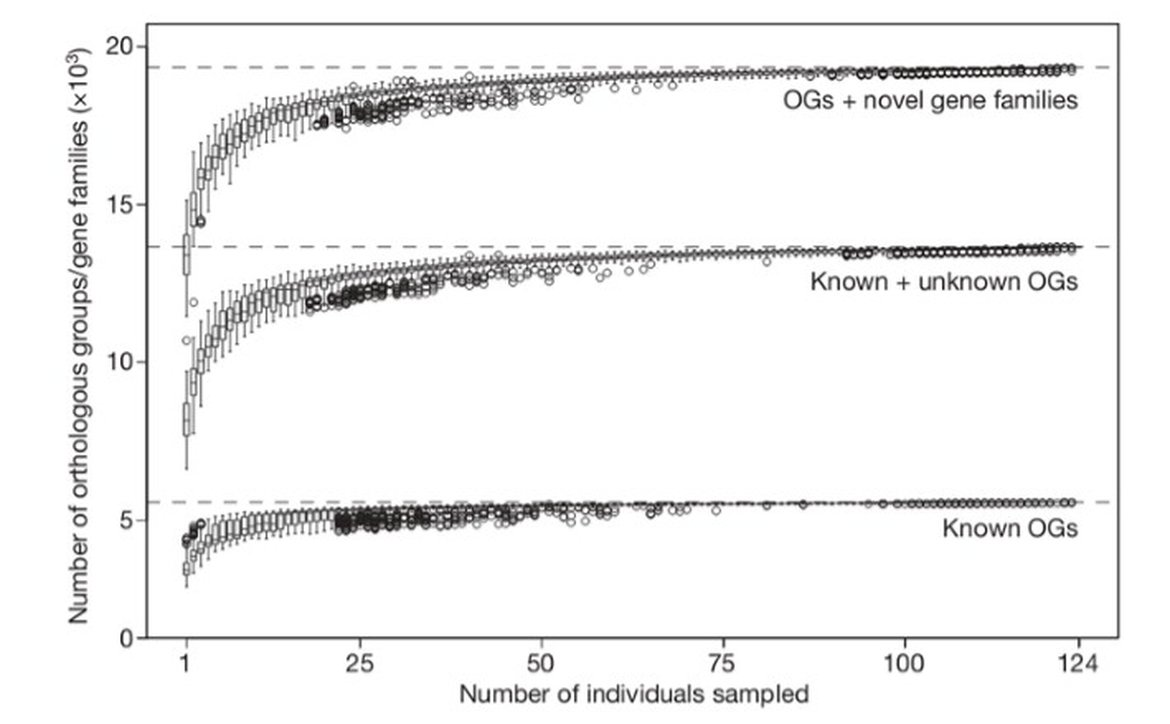
例图4、样本数目的增加使检测的功能信息趋于饱和
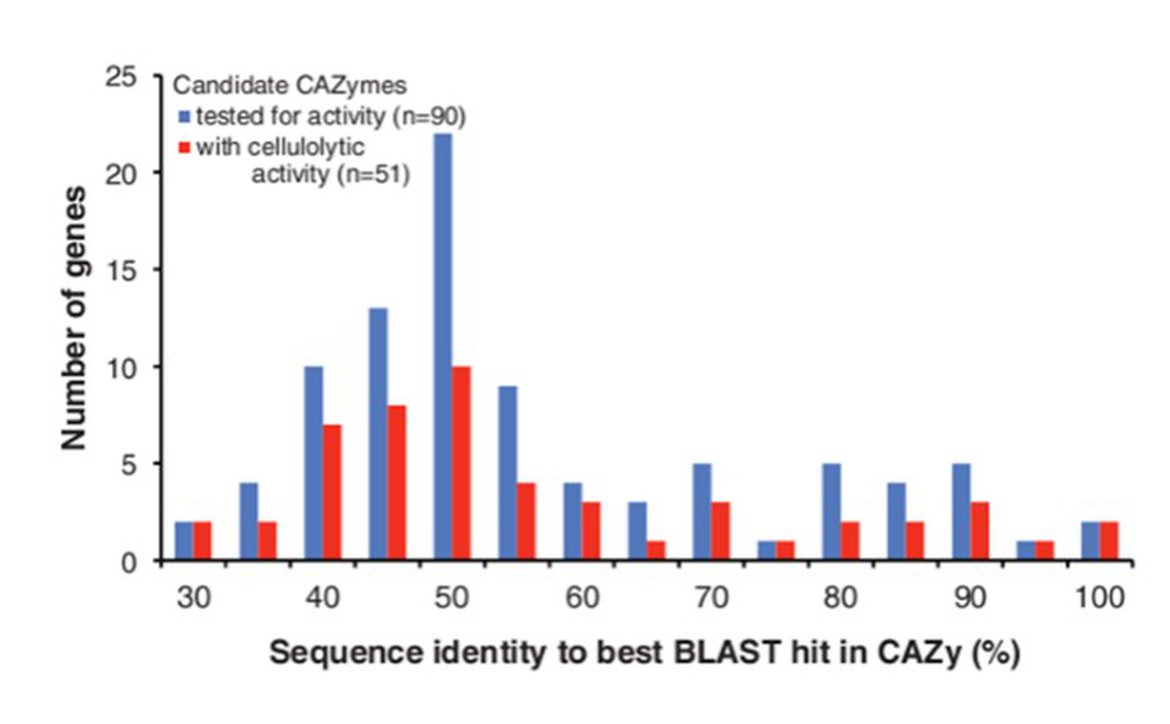
例图5、候选序列CAZymes活性检测统计
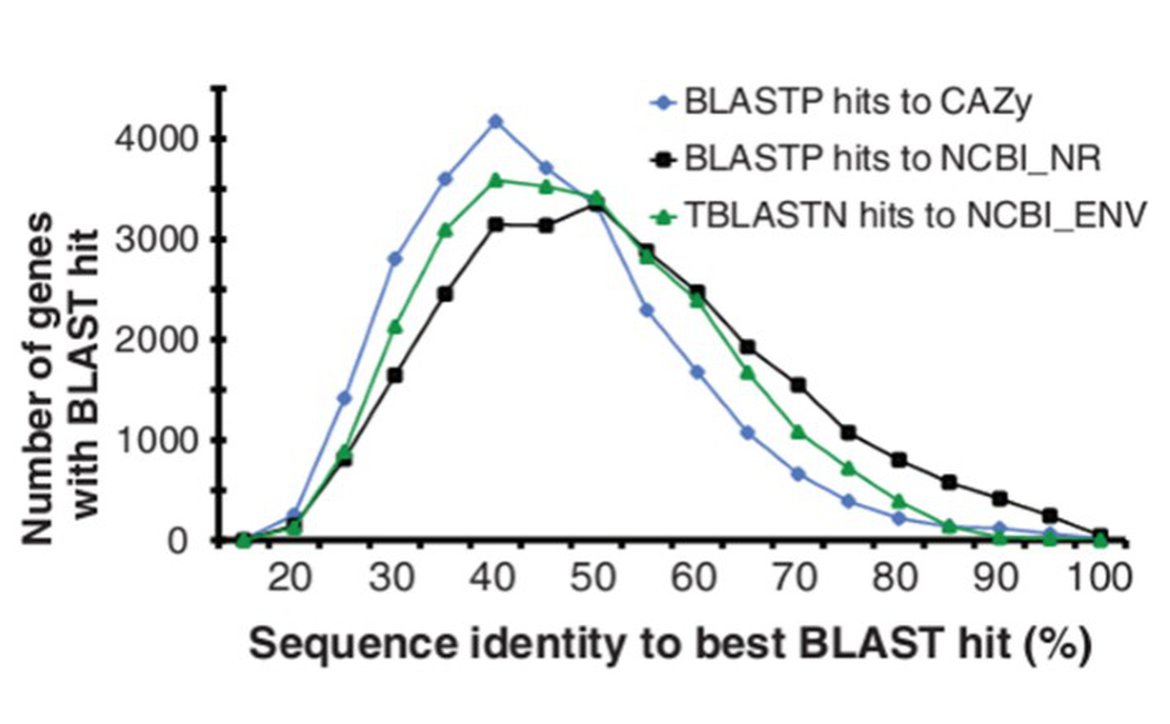
例图6、候选序列的功能分析及统计
参考文献
Segata N, Waldron L, Ballarini A, et al. Metagenomic microbial community profiling using unique clade-specific marker genes[J]. Nature Methods, 2012, 9(8): 811-814.
Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing[J]. Nature, 2010, 464(7285): 59-65.
Hess M, Sczyrba A, Egan R, et al. Metagenomic discovery of biomass-degrading genes and genomes from cow rumen[J]. Science, 2011, 331(6016): 463-467.